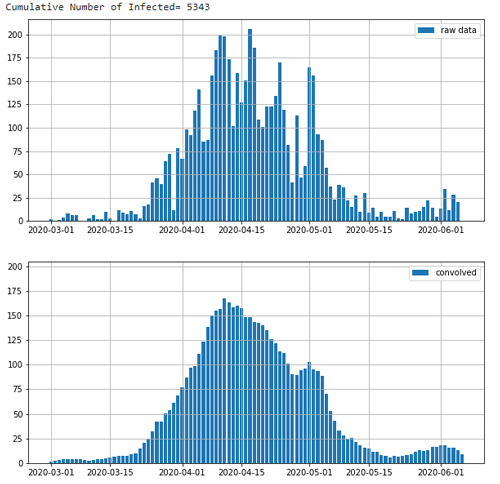
東京都新規感染者日変化と7日移動平均 -コロナウィルスとの闘い
Saturday, June 6th, 2020昨日(6月5日)までの東京都の公開データから新規感染者数の日変化と7日移動平均を上掲した。
上掲図では、3月1日から昨日までの日変化をプロットしている。図の作成は、東京都の公開データ(CSVデータ)からPythonを用いて作成している。Pythonスクリプトのソースを以下に添付している。
なお、図上部に、累積感染者数(Cumulative Number of Infected)として 5,3472人としているが、これは1月24日以降の累積。
Python Script source:
import matplotlib import matplotlib.pyplot as plt import pandas as pd import numpy as np import datetime df = pd.read_csv('https://stopcovid19.metro.tokyo.lg.jp/data/130001_tokyo_covid19_patients.csv') tokyo_df = df[['公表_年月日', '患者_年代', '患者_性別','退院済フラグ']] tokyo_df.columns = ['date', 'age', 'sex','recv'] tokyo_df_count = tokyo_df['date'].value_counts().sort_index(ascending=True) df_t = pd.DataFrame(tokyo_df_count) df_t.columns = ['infected'] dates = pd.date_range('20200124', 'today', freq='D') df = pd.DataFrame(pd.Series(range(len(dates)), index=dates)) df.columns = ['infected'] for dates in df.index: str_date = dates.strftime("%Y-%m-%d") try: df.infected[str_date] = df_t.infected[str_date] except: df.infected[str_date] = 0 #str_date='2020-05-28' #df.infected[str_date] = 15 list_infected = df.infected.to_list() date_index = [df.index[0].strftime('%Y-%m-%d')] for i in range(1, len(list_infected)): date_index.append(df.index[i].strftime('%Y-%m-%d')) x = pd.date_range(date_index[0], periods=len(date_index),freq='d') y = np.array(list_infected) y_cum = np.cumsum(y) print('Cumulative Number of Infected= ' + str(y_cum[len(y_cum)-1])) start_date = '2020-03-01' i_start = df.index.get_loc(start_date) num = 7 b = np.ones(num)/num y2 = np.convolve(y, b, mode='same') fig = plt.figure(figsize=(10,10)) x_d = x[i_start:] y_o = y[i_start:] y2_o= y2[i_start:] ax = fig.add_subplot(2,1,1) #ax = fig.add_axes([0,0,1,1]) ax.bar(x_d, y_o,label='raw data') #ax.bar(x, y_cum,label='raw data') ax.legend() ax.grid() ax = fig.add_subplot(2,1,2) #ax = fig.add_axes([0,0,1,1]) ax.bar(x_d, y2_o, label='convolved') ax.legend() ax.grid() plt.ylim(0, 205) plt.show()
.gif)